5-Keto-D-gluconate (ECMDB11731) (M2MDB000820)
| Record Information | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Version | 2.0 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Creation Date | 2012-05-31 14:20:36 -0600 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Update Date | 2015-09-17 15:41:46 -0600 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Secondary Accession Numbers |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Identification | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
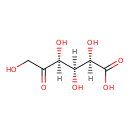
| Name: | 5-Keto-D-gluconate | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Description | 5-Keto-D-gluconate is metabolized from glucose in certain bacterial species. It is an intermediate in L-idonate degradation and ketogluconate metabolism. 5-Keto-D-gluconate 5-reductase catalyzes the reversible reduction of 5-ketogluconate to D-gluconate. This is the second reaction of the L-idonate catabolic pathway after uptake of L-idonate into the cell. The enzyme specifically reduces 5-ketogluconate using either NADH or NADPH. The enzyme is also specific for D-gluconate oxidation using NADP as the coenzyme, NAD does not serve as a coenzyme. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Structure | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Synonyms: |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Chemical Formula: | C6H10O7 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Weight: | Average: 194.1394 Monoisotopic: 194.042652674 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| InChI Key: | IZSRJDGCGRAUAR-UHFFFAOYSA-N | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| InChI: | InChI=1S/C6H10O7/c7-1-2(8)3(9)4(10)5(11)6(12)13/h3-5,7,9-11H,1H2,(H,12,13) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| CAS number: | 3470-36-8 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| IUPAC Name: | (2R,3S,4S)-2,3,4,6-tetrahydroxy-5-oxohexanoic acid | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Traditional IUPAC Name: | 5-dehydrogluconate | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| SMILES: | OCC(=O)C(O)C(O)C(O)C(O)=O | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Chemical Taxonomy | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Description | belongs to the class of organic compounds known as medium-chain hydroxy acids and derivatives. These are hydroxy acids with a 6 to 12 carbon atoms long side chain. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Kingdom | Organic compounds | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Super Class | Organic acids and derivatives | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Class | Hydroxy acids and derivatives | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Sub Class | Medium-chain hydroxy acids and derivatives | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Direct Parent | Medium-chain hydroxy acids and derivatives | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Alternative Parents |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Substituents |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Molecular Framework | Aliphatic acyclic compounds | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| External Descriptors |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Physical Properties | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| State: | Solid | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Charge: | -1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Melting point: | Not Available | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Experimental Properties: |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Predicted Properties |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Biological Properties | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Cellular Locations: | Cytoplasm | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reactions: | D-Altronate + NAD <> Hydrogen ion + NADH + 5-Keto-D-gluconate + D-Tagaturonate D-Galacturonate <> 5-Keto-D-gluconate 2,5-Diketo-D-gluconate + Hydrogen ion + NADH > 5-Keto-D-gluconate + NAD 2,5-Diketo-D-gluconate + Hydrogen ion + NADPH > 5-Keto-D-gluconate + NADP 5-Keto-D-gluconate + Hydrogen ion + NADPH <> Gluconic acid + NADP 5-Keto-D-gluconate + Hydrogen ion + NADH <> D-Galactonate + NAD 5-Keto-D-gluconate + Hydrogen ion + NADPH > D-Galactonate + NADP D-Galactonate + NAD > Hydrogen ion + NADH + 5-Keto-D-gluconate D-Altronate + NAD <> 5-Keto-D-gluconate + NADH + Hydrogen ion NAD(P)<sup>+</sup> + L-Idonate <> NAD(P)H + 5-Keto-D-gluconate + Hydrogen ion NAD(P)<sup>+</sup> + Gluconic acid <> NAD(P)H + 5-Keto-D-gluconate + Hydrogen ion Hydrogen ion + 2,5-Diketo-D-gluconate + NADPH <> 5-Keto-D-gluconate + NADP L-Idonate + NAD(P)(+) > 5-Keto-D-gluconate + NAD(P)H Gluconic acid + NAD(P)(+) > 5-Keto-D-gluconate + NAD(P)H Gluconic acid + NAD + NADP <> 5-Keto-D-gluconate + NADH + NADPH + Hydrogen ion L-Idonate + NAD + NADP <> 5-Keto-D-gluconate + NADH + NADPH + Hydrogen ion 2,5-Diketo-D-gluconate + NADPH + Hydrogen ion + NADPH > NADP + 5-Keto-D-gluconate + 5-Keto-D-gluconate L-Idonate + NADP > Hydrogen ion + NADPH + 5-Keto-D-gluconate + NADPH + 5-Keto-D-gluconate 5-Keto-D-gluconate + Hydrogen ion + NADPH + 5-Keto-D-gluconate + NADPH > NADP + Gluconic acid | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| SMPDB Pathways: |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| KEGG Pathways: |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| EcoCyc Pathways: |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Concentrations | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Not Available | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Spectra | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Spectra: |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| References | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| References: |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Synthesis Reference: | Not Available | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Material Safety Data Sheet (MSDS) | Not Available | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Links | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| External Links: |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Enzymes
- General function:
- Involved in oxidation-reduction process
- Specific function:
- D-altronate + NAD(+) = D-tagaturonate + NADH
- Gene Name:
- uxaB
- Uniprot ID:
- P0A6L7
- Molecular weight:
- 54808
Reactions
| D-altronate + NAD(+) = D-tagaturonate + NADH. |
- General function:
- Involved in glucuronate isomerase activity
- Specific function:
- D-glucuronate = D-fructuronate
- Gene Name:
- uxaC
- Uniprot ID:
- P0A8G3
- Molecular weight:
- 53987
Reactions
| D-glucuronate = D-fructuronate. |
| D-galacturonate = D-tagaturonate. |
- General function:
- Involved in oxidoreductase activity
- Specific function:
- Catalyzes a reversible reduction of 5-ketoglutanate to form D-gluconate. Dependent on NADP, almost inactive with NAD
- Gene Name:
- idnO
- Uniprot ID:
- P0A9P9
- Molecular weight:
- 27563
Reactions
| D-gluconate + NAD(P)(+) = 5-dehydro-D-gluconate + NAD(P)H. |
- General function:
- Involved in oxidoreductase activity, acting on the CH-OH group of donors, NAD or NADP as acceptor
- Specific function:
- Catalyzes the NADPH-dependent reduction of glyoxylate and hydroxypyruvate into glycolate and glycerate, respectively. Can also reduce 2,5-diketo-D-gluconate (25DKG) to 5-keto-D- gluconate (5KDG), 2-keto-D-gluconate (2KDG) to D-gluconate, and 2- keto-L-gulonate (2KLG) to L-idonate (IA), but it is not its physiological function. Inactive towards 2-oxoglutarate, oxaloacetate, pyruvate, 5-keto-D-gluconate, D-fructose and L- sorbose. Activity with NAD is very low
- Gene Name:
- ghrB
- Uniprot ID:
- P37666
- Molecular weight:
- 35395
Reactions
| Glycolate + NADP(+) = glyoxylate + NADPH. |
| D-glycerate + NAD(P)(+) = hydroxypyruvate + NAD(P)H. |
| D-gluconate + NADP(+) = 2-dehydro-D-gluconate + NADPH. |
- General function:
- Involved in zinc ion binding
- Specific function:
- Catalyzes the NADH/NADPH-dependent oxidation of L- idonate to 5-ketogluconate (5KG)
- Gene Name:
- idnD
- Uniprot ID:
- P39346
- Molecular weight:
- 37146
Reactions
| L-idonate + NAD(P)(+) = 5-dehydrogluconate + NAD(P)H. |
- General function:
- Involved in zinc ion binding
- Specific function:
- Putative L-galactonate oxidoreductase that is required for growth on L-galactonate as the sole carbon source
- Gene Name:
- yjjN
- Uniprot ID:
- P39400
- Molecular weight:
- 36448
Transporters
- General function:
- Involved in gluconate transmembrane transporter activity
- Specific function:
- Transports L-idonate, D-gluconate and 5-keto-D- gluconate, from the periplasm across the inner membrane
- Gene Name:
- idnT
- Uniprot ID:
- P39344
- Molecular weight:
- 46041
- General function:
- Involved in transporter activity
- Specific function:
- Non-specific porin
- Gene Name:
- ompN
- Uniprot ID:
- P77747
- Molecular weight:
- 41220
- General function:
- Involved in transporter activity
- Specific function:
- Uptake of inorganic phosphate, phosphorylated compounds, and some other negatively charged solutes
- Gene Name:
- phoE
- Uniprot ID:
- P02932
- Molecular weight:
- 38922
- General function:
- Involved in transporter activity
- Specific function:
- OmpF is a porin that forms passive diffusion pores which allow small molecular weight hydrophilic materials across the outer membrane. It is also a receptor for the bacteriophage T2
- Gene Name:
- ompF
- Uniprot ID:
- P02931
- Molecular weight:
- 39333
- General function:
- Involved in transporter activity
- Specific function:
- Forms passive diffusion pores which allow small molecular weight hydrophilic materials across the outer membrane
- Gene Name:
- ompC
- Uniprot ID:
- P06996
- Molecular weight:
- 40368